腸道微生物與宿主之間互惠共生,密不可分,定植在宿主體內的共生微生物可以透過多種機制影響宿主生理與病理生理功能。其中, 膽汁酸是溝通宿主與腸道微生物的關鍵信使分子 【1-3】 。 人體在肝臟由至少17種不同的酶將膽固醇代謝為膽酸(CA)和鵝去氧膽酸(CDCA)等初級膽汁酸,而初級膽汁酸透過膽汁釋放到腸道,經由腸道微生物代謝為去氧膽酸(DCA)、石膽酸(LCA)等次級膽汁酸。次級膽汁酸可以透過調控法尼醇X受體(FXR)、膽汁酸G蛋白偶聯受體5 (TGR5)、維生素D受體 (VDR) 等宿主受體,在宿主糖脂代謝、免疫應答等多個生理與病理生理過程中發揮關鍵作用 【4-8】 ,因而受到了研究人員的廣泛關註。然而, 目前腸道微生物對膽汁酸的修飾類別還有待挖掘,不同膽汁酸與疾病調控的分子機制尚不明確 。此外,對菌群膽汁酸代謝的生物合成通路解析是理解次級膽汁酸生成機制、開發靶向性調控策略的必經之路,但目前僅有幾類次級膽汁酸的合成通路得到解析 【9,10】 ,很難透過常規的組學手段對新型膽汁酸合成通路進行挖掘, 如何不基於先驗知識發現新型膽汁酸的生物合成通路仍然是一項非常有挑戰性的學科問題。
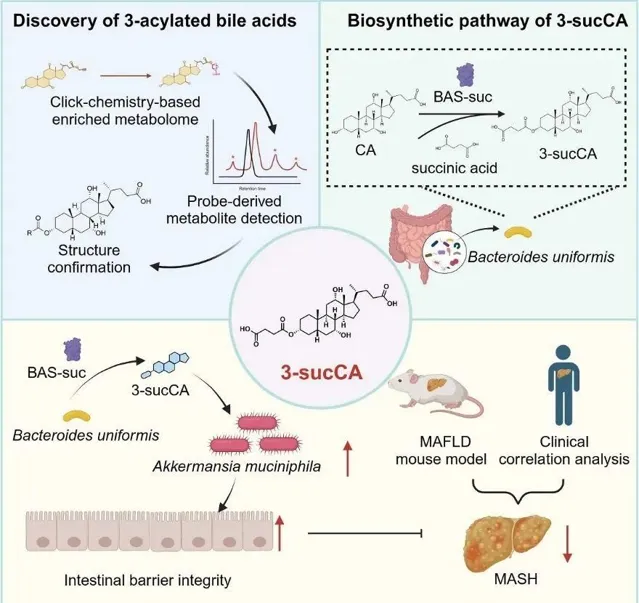
2024年4月22日,北京大學醫學部基礎醫學院/北京大學第三醫院醫學創新研究院 姜長濤 教授團隊、北京大學第三醫院 喬傑 院士團隊、北京大學第三醫院 龐艷莉 團隊、溫州醫科大學附屬第一醫院 鄭明華 團隊以及北京大學醫學部藥學院 賈彥興 團隊合作,在 Cell 雜誌線上發表了題為 Gut symbionts alleviate MASH through a secondary bile acid biosynthetic pathway 的研究論文,構建了基於點選化學富集策略結合非靶向代謝組學方法的次級膽汁酸挖掘體系,發現腸道菌群對膽汁酸的全新修飾類別—3-酰基化修飾,並在菌群、小鼠、人群三個維度系統地檢測了7種3-酰基化膽汁酸的豐度及流行率。透過分離培養建構腸道菌株庫及大規模篩選,研究團隊發現單形擬桿菌( Bacteroides uniformis )是 3-琥珀酰膽酸 ( 3-sucCA )主要產生者。進一步,透過基於活性的蛋白追蹤純化策略,研究團隊挖掘出 3-sucCA的合成酶 ——「 BAS-suc 」。最後,臨床佇列提示3-sucCA與 代謝相關脂肪性肝炎 ( MASH )行程呈顯著的負相關性。研究團隊深入解析了3-sucCA對MASH行程的改善作用與分子機制,發現其透過菌群重塑—促進益生菌嗜黏蛋白阿克曼氏菌( Akkermansia muciniphila )的生長,改善腸屏障損傷,降低慢性低水平炎癥,從而逆轉小鼠的MASH行程。
姜長濤教授團隊長期致力於代謝性疾病的發病機制與轉化醫學研究,重點聚焦腸道菌群介導的器官間對話。團隊以腸道菌群產生的多種代謝酶為切入點及潛在幹預靶點,首創「腸道菌源酶跨物種調控宿主穩態」新理論:提出腸道菌源宿主同工酶新概念,發現其廣泛存在且能跨物種調控宿主疾病,揭示菌源DPP4損傷宿主血糖穩態、介導臨床降糖藥物響應性的分子機制( Science 2023);揭示吸煙期間尼古丁在腸道的大量蓄積,透過腸AMPK—神經醯胺軸加重MASH的新機制。首次發現降解尼古丁的腸道共生菌,提出腸道共生菌透過新型尼古丁降解酶NicX改善代謝性疾病的新幹預策略( Nature 2022)。此外,研究團隊積累了大量關於腸道菌源膽汁酸與代謝性疾病的前期工作,揭示了腸道菌群透過多種膽汁酸代謝酶生成GUDCA、GDCA等膽汁酸,介導菌群—器官互作,調控機體代謝與免疫,靶向膽汁酸代謝酶,能夠改善代謝性疾病( Nature Microbiol ogy 2024; Cell Metabolism 2021a, b; Nature Medicine 2018, 2019; Journal of Clinical Investigation 2021, 2015),以上發現揭示了腸道菌源膽汁酸等是介導器官間對話的關鍵介質。
除了廣泛研究的傳統膽汁酸,是否還有其他未知的新型菌源膽汁酸可以參與到代謝性疾病中的調控作用中?近期科研人員透過反向代謝組等方法發現了膽汁酸的24位元羧基修飾 【11】 。然而,由於糞便成分復雜、膽汁酸質譜響應性較低,基於單純代謝組學策略的新型膽汁酸挖掘存在一定局限。那麽,是否有高效的菌源膽汁酸挖掘體系,既可以富集膽汁酸衍生物,又可以進行快速辨別?除了已經廣泛發現的24位元羧基修飾的新型菌源膽汁酸外 【12-14】 ,是否存在其他官能團上進行修飾的菌源膽汁酸?因此,開發全新的膽汁酸挖掘體系並研究其生物合成通路及生理作用具有重要意義。
研究團隊基於點選化學的原理,建立了一套菌源膽汁酸挖掘體系。該體系包含能發生點選化學反應的帶有炔基的膽酸探針以及帶有疊氮基的ACER·resim。膽酸探針經過健康誌願者糞便菌群代謝轉化後,其衍生物透過點選化學反應以及ACER·resim的富集,得到濃縮膽汁酸衍生物而含有較少無關代謝物的富集液。膽汁酸衍生物的點選化學產物在非靶向代謝組中展現出100.0761的特征峰,因而實作了膽汁酸衍生物的高效富集和靈敏辨別。透過分子網絡分析,研究團隊發現了一系列包含酰基化的新型菌源膽汁酸,包括:甲酰化、乙酰化、丙酰化、丁酰化、乙醇酰化、丙二酰化、以及琥珀酰化。透過大規模糞便菌群發酵液分離與化學合成比對,研究團隊在膽酸所包含的3個可發生酰基化反應的羥基位點(3位、7位、12位元)中,確認所有新型菌源膽汁酸的酰基化位置都發生在3位上。最後,研究團隊在菌群、小鼠、人群三個維度系統地檢測了新型膽汁酸的豐度及流行率,這些酰基化膽汁酸廣泛存在,可能存在一定的生理作用。
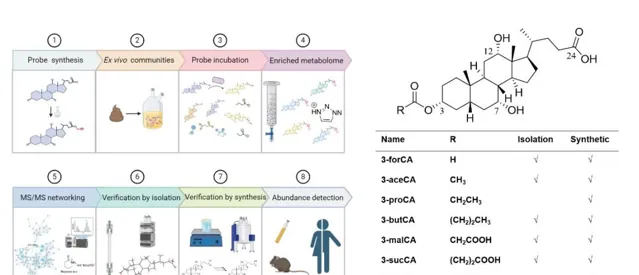
圖1. 菌源膽汁酸挖掘體系與3-酰基化膽汁酸結構
在評估新型膽汁酸的豐度及流行率過程中,3-琥珀酰膽酸(3-sucCA)在健康誌願者糞便樣本中表現出了最高的豐度與流行率。為探索3-sucCA的主要腸道菌來源,研究團隊收集了3-sucCA豐度最高的健康誌願者的糞便樣本,透過分離培養建構腸道菌株庫、大規模菌株篩選、小鼠定植檢測實驗確認了單形擬桿菌在體外或體內都存在3-sucCA合成能力。
生物合成通路的探索是菌源膽汁酸研究領域的重點和難點 【10, 15-17】 ,也是精細調控腸道菌群作用的關鍵一步。為探索單形擬桿菌中負責3-sucCA生物合成的關鍵酶,研究團隊透過大腸桿菌過表達實驗檢驗了單形擬桿菌中所有的16種註釋為酰基轉移酶的基因,然而這些候選基因都沒有產3-sucCA的活性。接下來,研究團隊以單形擬桿菌裂解液粗酶反應為基礎,透過基於活性的蛋白純化與鑒定方法,逐步根據裂解液 > 80%硫酸銨蛋白沈澱 > 疏水相互作用色譜(HIC)> 離子交換色譜(IEC)層層純化,最終利用蛋白質譜分析發現了共118候選蛋白。團隊利用蛋白異源表達實驗將以上118個蛋白全部進行一一驗證,發現只有當過表達一個被註釋為β-內醯胺酶的蛋白A7UYF6時可以使大腸桿菌獲得產生3-sucCA的能力。進一步地,研究團隊透過基於CRISPR-Cas系統的厭氧菌基因剔除實驗、近緣同屬菌株過表達實驗、以及純酶體外酶促實驗驗證了該酶負責3-sucCA的生物合成,並將該酶命名為「BAS-suc」。
3-sucCA如何促進嗜黏蛋白阿克曼氏菌生長?轉錄組與非靶向代謝組揭示出3-sucCA可以透過促進嗜黏蛋白阿克曼氏菌對葡萄糖的胺化與利用進而加速其細胞壁主要成分—肽聚糖的合成。不同碳源受質利用實驗也說明:當胺基糖匱乏,葡萄糖作為唯一碳源情況下,3-sucCA對嗜黏蛋白阿克曼氏菌存在最大的生長加速作用。進一步地,表面等離子共振技術(SPR)揭示出嗜黏蛋白阿克曼氏菌唯一可以實作胺化/脫胺的酶NagB與3-sucCA存在較強的相互作用。由於嗜黏蛋白阿克曼氏菌缺乏高效的遺產操作體系,研究團隊在大腸桿菌中透過基因剔除與替換,成功地模擬了嗜黏蛋白阿克曼氏菌苛刻的營養需求,並行現3-sucCA透過NagB實作了對嗜黏蛋白阿克曼氏菌生長的調控作用。
以上結果在臨床佇列中也得到驗證,具體表現為:單形擬桿菌與嗜黏蛋白阿克曼氏菌豐度隨著MAFLD疾病行程逐漸下降,並且 bas-suc 基因豐度與3-sucCA和嗜黏蛋白阿克曼氏菌顯著正相關,而與MAFLD疾病指標顯著負相關。這些結果揭示了3-sucCA作為新型菌源膽汁酸透過菌群互作改善MAFLD的新範式。
膽汁酸作為「腸肝迴圈」的重要介質,是 宿主與菌群協同代謝的經典範例,也是腸道菌群調控宿主生理活動的關鍵信使。菌源膽汁酸結構存在多樣性,但其種類不清、生理功能不明、合成通路有待挖掘,缺少特異性挖掘體系。本工作構建了一種全新的基於點選化學的菌源膽汁酸挖掘體系,並行現一大類之前未被表征的新型菌源膽汁酸—3-酰基化膽酸,並深入地探索了3-sucCA透過腸道菌群互作對MASH進展的改善作用與分子機制,為未來臨床治療MASH等代謝性疾病的潛在藥物開發提供了研究基礎;此外,本工作系統解析了3-sucCA的生物合成通路,並行現了一類全新的膽汁酸代謝酶BAS-suc,為菌源膽汁酸的生物合成研究提供了新的方向,同時也為調控疾病的腸道菌源酶挖掘研究提供了新範式。
姜長濤教授、喬傑院士、龐艷莉研究員、鄭明華教授與賈彥興教授為本文的共同通訊作者。北京大學醫學部基礎醫學院博士後聶啟興、博士研究生羅茜、汪鍇副研究員、博士後丁勇以及藥學院博士研究生賈淑密為本文的共同第一作者。
姜長濤,北京大學長聘教授、博雅特聘教授,基礎醫學院副院長,免疫學系主任,國家傑出青年科學基金獲得者、科學探索獎獲得者。從事腸道共生菌與代謝性疾病的研究。首創「腸道菌源酶跨物種調控宿主穩態」新理論:提出菌源宿主同工酶的新概念,揭示菌源DPP4介導臨床降糖藥物響應性的分子機制,為靶向腸道菌群精準幹預疾病開辟了全新方向;發現菌群—宿主互作的關鍵信使—膽汁酸的全新菌源修飾類別及生物合成通路,是脂肪性肝炎防治的新策略;首次發現了降解尼古丁的腸道共生菌及其在脂肪性肝炎發病中的關鍵保護作用。近5年在 Cell (2024)、 Science (2023)、 Nature (2022)、 Nature Microbiology (2024)、 Nature Cardiovascular Research (2023)、 Cell Metabolism (2021a, 2021b, 2019)、 Nature Medicine (2019)等雜誌發表SCI論文二十余篇。現因研究發展需要,誠招聘博士後。受聘者具有良好溝通合作能力和團隊精神,具備獨立從事科研工作的能力,有良好的英語表達和論文寫作能力;博士期間以第一作者或共同第一作者發表過高影響因子SCI論文者優先;具有生物合成、受體、訊息傳遞、免疫、代謝等相關領域的研究背景者優先;已取得或即將取得博士學位,年齡不超過35周歲。
有意應聘者請按下述流程應聘:
• 應聘者請準備個人簡歷等相關材料進行發送,應聘理由請註明「博士後應聘+姓名」;
• 招聘長期有效。
參考文獻
[1] PERINO A, SCHOONJANS K. Metabolic Messengers: bile acids [J]. Nature metabolism , 2022, 4(4): 416-23.
[2] COLLINS S L, STINE J G, BISANZ J E, et al. Bile acids and the gut microbiota: metabolic interactions and impacts on disease [J]. Nature reviews Microbiology , 2023, 21(4): 236-47.
[3] FUCHS C D, TRAUNER M. Role of bile acids and their receptors in gastrointestinal and hepatic pathophysiology [J]. Nature reviews Gastroenterology & hepatology , 2022, 19(7): 432-50.
[4] SUN L, XIE C, WANG G, et al. Gut microbiota and intestinal FXR mediate the clinical benefits of metformin [J]. Nature medicine , 2018, 24(12): 1919-29.
[5] ZHENG X, CHEN T, JIANG R, et al. Hyocholic acid species improve glucose homeostasis through a distinct TGR5 and FXR signaling mechanism [J]. Cell metabolism , 2021, 33(4): 791-803.e7.
[6] HANG S, PAIK D, YAO L, et al. Bile acid metabolites control T(H)17 and T(reg) cell differentiation [J]. Nature , 2019, 576(7785): 143-8.
[7] SONG X, SUN X, OH S F, et al. Microbial bile acid metabolites modulate gut RORγ(+) regulatory T cell homeostasis [J]. Nature , 2020, 577(7790): 410-5.
[8] CAMPBELL C, MCKENNEY P T, KONSTANTINOVSKY D, et al. Bacterial metabolism of bile acids promotes generation of peripheral regulatory T cells [J]. Nature , 2020, 581(7809): 475-9.
[9] RIDLON J M, GASKINS H R. Another renaissance for bile acid gastrointestinal microbiology [J]. , 2024.
[10] FUNABASHI M, GROVE T L, WANG M, et al. A metabolic pathway for bile acid dehydroxylation by the gut microbiome [J]. Nature , 2020, 582(7813): 566-70.
[11] GENTRY E C, COLLINS S L, PANITCHPAKDI M, et al. Reverse metabolomics for the discovery of chemical structures from humans [J]. Nature , 2024, 626(7998): 419-26.
[12] QUINN R A, MELNIK A V, VRBANAC A, et al. Global chemical effects of the microbiome include new bile-acid conjugations [J]. Nature , 2020, 579(7797): 123-9.
[13] MULLOWNEY M W, FIEBIG A, SCHNIZLEIN M K, et al. Microbially catalyzed conjugation of GABA and tyramine to bile acids [J]. Journal of bacteriology , 2024, 206(1): e0042623.
[14] MOHANTY I, MANNOCHIO-RUSSO H, SCHWEER J V, et al. The underappreciated diversity of bile acid modifications [J]. Cell , 2024, 187(7): 1801-18.e20.
[15] GUZIOR D V, OKROS M, SHIVEL M, et al. Bile salt hydrolase acyltransferase activity expands bile acid diversity [J]. Nature , 2024, 626(8000): 852-8.
[16] RIMAL B, COLLINS S L, TANES C E, et al. Bile salt hydrolase catalyses formation of amine-conjugated bile acids [J]. Nature , 2024, 626(8000): 859-63.
[17] SATO Y, ATARASHI K, PLICHTA D R, et al. Novel bile acid biosynthetic pathways are enriched in the microbiome of centenarians [J]. Nature , 2021, 599(7885): 458-64.
[18] CANI P D, DEPOMMIER C, DERRIEN M, et al. Akkermansia muciniphila: paradigm for next-generation beneficial microorganisms [J]. , 2022, 19(10): 625-37.
[19] HAN Y, LI L, WANG B. Role of Akkermansia muciniphila in the development of nonalcoholic fatty liver disease: current knowledge and perspectives [J]. Frontiers of medicine , 2022, 16(5): 667-85.
[20] MAIER L, PRUTEANU M, KUHN M, et al. Extensive impact of non-antibiotic drugs on human gut bacteria [J]. Nature , 2018, 555(7698): 623-8.













